Transkriptionelle Regulierung der Lignin-Biosynthese in Miscanthus
Die C4-Pflanze Miscanthus erbringt einen hohen Biomasseertrag, selbst auf marginalen Böden, und ist daher ein wichtiger Kandidat als zukünftiger Biomasseproduzent. Beim Einsatz pflanzlicher Biomasse für unterschiedliche Anwendungen ist deren Zusammensetzung von zentraler Bedeutung. Hauptbestandteile der pflanzlichen Zellwand sind die Polymere Zellulose, Hemizellulose und Lignin. Dabei ist die Lignin-Komponente von besonderem Interesse, da sie den Zugang zu den Zuckerkomponenten behindert, und daher in Bioraffinerie-Konzepten abgetrennt werden muss. Andererseits könnte Lignin selbst Ausgangsbasis für aromatische Plattformchemikalien sein, wenn es zukünftig gelingt, funktionalisiertes Lignin mit definierten Eigenschaften herzustellen. In diesem Kontext ist die Züchtung von Miscanthus-Pflanzen mit erhöhtem oder erniedrigtem Lignin-Gehalt bzw. geänderter Lignin-Zusammensetzung von großem Interesse.
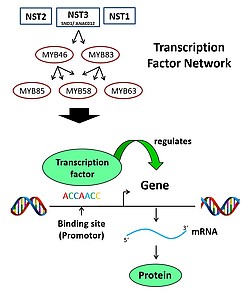
Ziel des Teilprojektes war es daher, die transkriptionelle Regulation der Lignin-Biosynthese in Miscanthus sinensis aufzuklären. Letztere erfolgt über ein komplexes Netzwerk an Transkriptionsfaktoren (TF), die von verschiedenen Anzuchtparametern moduliert werden. Ausgehend von Sequenzinformation aus Arabidopsis, Mais und Hirse wurden Lignin-Biosynthese steuernde TF für M. sinensis identifiziert (RNAseq-Datenbank). Dabei wurden Vertreter aller drei die Lignin-Biosynthese beeinflussenden Regulationsebenen berücksichtigt (Ebene 3: SND1, Master Regulator für sekundäre Zellwand; Ebene 2: MYB46/MYB83; Ebene 1: MYB85/MYB58/MYB63 - Genbezeichnungen entsprechend der Arabidopsis Nomenklatur), aber auch zwei reprimierende TF (MsMYB31 und MsMYB42). Der Zellwandregulator MsSND1 wurde hinsichtlich seiner Funktionalität näher charakterisiert. So konnte die Arabidopsis Doppelmutante nst1/nst3 mit dem MsSND1-Gen komplementiert werden, und unter Einsatz eines DEX-induzierbaren MsSND1-Genkonstrukts gelang die Identifizierung direkter und indirekter Ziel-Gene.
Durch partielle Genomsequenzierung wurde für M. sinensis eine zusätzliche Datenbank geschaffen, die die Klonierung von Promotorsequenzen sowohl der TF-Gene als auch der Lignin-Biosynthese-Gene erlaubt. Durch transiente Expression in Tabakblättern und durch die Messung der Promotor-aktivierenden Wirkung konnte gezeigt werden, dass alle bioinformatisch identifizierten TF-Kandidaten die Promotoren der vorausgesagten Ziel-Gene der Lignin-Biosynthese aktivieren, darunter auch den Promoter des Laccase-Gens MsLAC1, ein Enzym, über dessen veränderte Expression Lignin-Menge und –Zusammensetzung beeinflusst werden können. In Zusammenarbeit mit der Arbeitsgruppe von Shawn Mansfield (UBC, Vancouver) konnte der Einfluss dieser TFs auf die qualitative Lignin-Zusammensetzung (S-/G-Lignin etc.) nachgewiesen werden.
| Projekttitel | Multifaktorielle Regulation der Ligninbiosynthese: Ein Schlüsselfaktor für Lignozellulose-basierte Wertschöpfungsketten in der Bioökonomie |
| Institution | |
| Beteiligte Wissenschaftler | Prof. Thomas Rausch, Dr. Sebastian Wolf, Philippe Golfier, Feng He, Wan Zhang |
| Projektstatus | abgeschlossen |
